bozza di scritto completo in italiano
full draft article in italian
modello di calcolo
Funzione logistica per
|
|
Bruno Caudana
b.caudana@adaptive.it
http://www.adaptive.it
16-mar-2020
|
"Non importa quanto bella sia la tua teoria, non importa quanto tu sia intelligente. |
| modello+fitting | stime $R_{0}$ | lockdown | COVID-mondo |
| nota evoluzion | nota su mortalità |
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
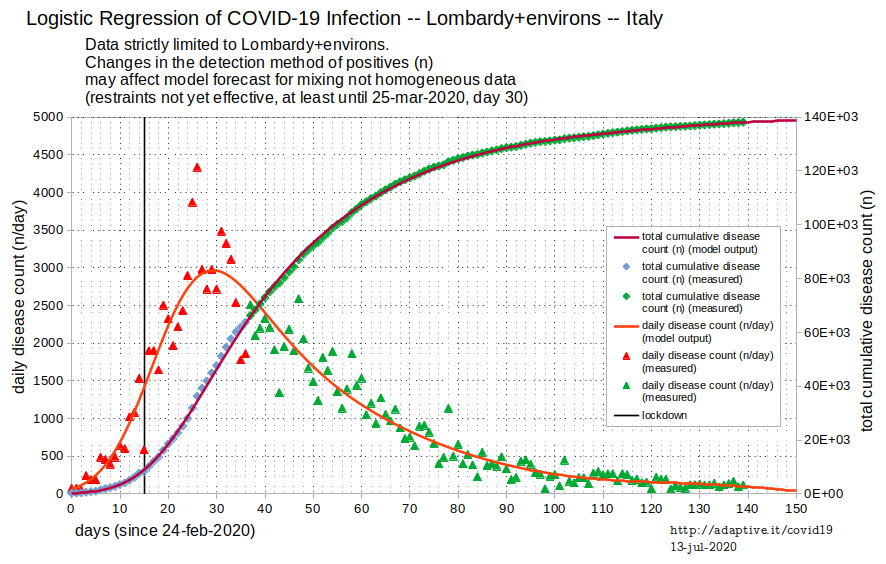
La curva studiata è quella dei casi totali cumulati della patologia macroscopica, secondo la definizione del Ministero della Salute.
Va fatto notare che, con il procedere dell'epidemia, si sono andati sommando come nuovi casi gli accertamenti positivi fatti allo stesso soggetto più volte. Questo sovrastima il reale numero di casi totali.
Inoltre, non è stato adottato un metodo uniforme per il riconoscimento dei casi di patologia macroscopica, il che porta a distorsioni dell'insieme di dati.
Ma questo è il data set disponibile. Un data set depurato da queste distorsioni avrebbe consentito un fitting più accurato e calzante sul fenomeno.
In una popolazione, quanti si ammalano di patologia macroscopica durante una epidemia dipende da:
L'andamento temporale dei casi cumulati di patologia macroscopica contiene in modo aggregato questa informazione incognita. Non è semplice disaggregare questa informazione. Forse impossibile.
Ma non si può dire che chi NON HA SVILUPPATO anticorpi specifici per SARS-CoV-2 è automaticamente suscettibille ad infettarsi. Questa è una semplificazione prudenziale, ma troppo conservativa. Implica assumere arbitrariamente che tutta una popolazione sia interamente suscettibile al virus.
Questa assunzione è molto improbabile e in contrasto con i meccanismi evoluzionistici che hanno forgiato il sistema immunitario degli ospiti: improbabile che un patogeno non sia attaccabile in più bersagli biochimici, alcuni dei quali già riconosciuti e memorizzati dal sistema immunitario.
(update)
Di fatto non si sa quale è la suscettibilità iniziale di una popolazione ad un pur nuovo virus. Potrebbero agire fattori di protezione genetica o indotta da pregressa storia immunitaria soggettiva. Per esempio: questo virus potrebbe avere bersagli biochimici comuni ad altri Corona Virus già riconosciuti dallo spettro immunitario della popolazione ospite. Inoltre, storicamente, non si è mai visto una epidemia infettare una popolazione completa.
Quanti muoiono o guariscono dipende da:
Una epidemia in evoluzione libera, semplificando all'osso, ha una fase di accensione, raggiunge un picco, infine si spegne in modo simmetrico a come è iniziata. Questa è la cornice del modello matematico adottato.
La curva effettiva misurata mostra di seguire un andamento di accensione e spegnimento senza segni evidenti e inequivoci dell'effetto del contrasto alla diffusione virale rispetto alla evoluzione libera dell'epidemia
La curva presenta alta impennata di salita, con presumibile ritardo di fase della patologia macroscopica rispetto alla diffusione virale; un ulteriore ritardo di fase si ha sull'andamento dei morti e dei dichiarati "guariti". Questo potrebbe indicare che il virus è già endemico alla data del lockdown, almeno nella zona lombarda e aree limitrofe.
Con l'evolvere dell'epidemia, sono aumentati i tamponi usati per rintracciare in modo più aggressivo i positivi al virus, rispetto ad inizio epidemia. Questo presumibilmente causa una apparente diminuzione di aggressività del virus perché vengono diagnosticati più positivi con sintomi più lievi, rispetto ad inizio epidemia. Inoltre, nel frattempo si è imparato a curare in modo più efficace e si comincia a manifestare resistenza immunitaria nella popolazione ospite. Non è indebolita la aggressività del virus, ma è rafforzata la resistenza complessiva della popolazione ospite.
Inoltre, questo è sicuramente un fattore cha causa l'andamento asimmetrico dello spegnimento dell'epidemia mostrato dalla curva. Un altro fattore può essere un effetto dei mutati atteggiamenti relazionali per non infettarsi (buon senso nel distanziarsi, più che lockdown normativo). Altri fattori incogniti di secondo livello possono intervenire a modulare la non linearità di diffusione virale (es: effetti secondari in aumento o diminuzione dovute alle variazioni di comportamento, ec.).
Comunque, di fronte a una minaccia grave e sconosciuta, meglio abbondare con le precauzioni. Questo modello non considera in alcun modo l'eventuale decadimento delle difese immunitarie acquisite dalla popolazione: se le difese immunitarie decadono rapidamente, allora l'infezione può ricomparire altrettanto rapidamente. Vedremo...!
Il modello adottato per la stima/fitting dei casi totali cumulati $f(t)$ è una estensione dell'equazione differenziale ridotta di Verhulst: $$\dot W(t) = \beta W(t)(\Omega -W(t))$$ Essa rappresenta un fenomeno di diffusione in ambiente chiuso a risorse limitate, dove ciò che determina la diffusione determina anche una resistenza crescente alla diffusione stessa per effetto del processo di diffusione. Essa ha come soluzione $W(t)=\Omega {\frac {1}{1+e^{-(\beta_{0} + \beta_{1}t )}}} $, cioè la curva detta a sigmoide o logistica.
L'equazione differenziale di Verhulst è qui adottata come cornice a rappresentare un fenomeno di diffusione in ambiente a risorse limitate, quale una epidemia è.
L'estensione si ispira all'approccio adottato nella statistica di Fermi-Dirac, dove il fattore di proporzionalità $\beta$ è sostituito da una funzione $\beta(t)$ più complessa. Inoltre, al posto di $ \Omega $ (il $c_{t}^{\ast}=p_{rif} \cdot i_{c}^{\ast}$ nel modello epidemico), viene adottato il suo andamento temporale $ \Omega(t) $ stimato in corsa, a tener conto del fatto che una epidemia si espande anche spazialmente. La popolazione di riferimento considerata è quella di
Il modello fisico-matematico specifico adottato per la stima/fitting dei casi totali cumulati $f(t)$, con i conseguenti casi giornalieri $f_{}^{\prime}(t)$, è espresso dalle seguenti formule: $$f(t)= c_{t}^{\ast} {\frac {1}{1+e^{-\beta(t)}}} \, , \qquad f_{}^{\prime}(t)= c_{t}^{\ast} \frac{ \frac{\mathrm{d}}{\mathrm{d}t}\beta(t) \cdot e^{-\beta(t) } } {\left( 1+e^{-\beta(t)} \right)^2} $$ dove $\beta(t)$ è la funzione che determina il fattore $\beta$ dell'equazione differenziale di Verhulst.
Il fitting non lineare del parametro indice di contagio ($i_{c}^{\ast}= c_{t}^{\ast} / p_{rif}$) -- nonché fattore di normalizzazione per la trasformata logistica -- è ottenuto mediante algoritmo evoluzionistico DEPS (Differential Evolution and Particle Swarm Optimization) minimizzando l'errore quadratico del modello rispetto ai dati originali, non scalati secondo logistica. In particolare, viene minimizzato l'errore quadratico generato dai casi giornalieri rispetto a $f_{}^{\prime}(t)$, presi ciascuno in scala logaritmica, soggetto al vincolo $z=f_{}^{\prime}(t) > 0$ per i casi giornalieri ricalcolati dal modello.
In formule: $$ err_{m} = \sqrt{\frac{1}{N}\sum_{k=1}^{N}(\ln(n_{k})- \ln(\hat{z}_{k}))^{2}} \quad , \qquad \sigma_{min} = \min(err_{m}) $$ col vincolo $\hat{z}_{k} > 0$ per ogni conteggio ricalcolato dal modello.
I dettagli del metodo sono nello scritto esteso.
Al momento ho scelto un polinomio in $t$, $P(t)= \beta_{0} + \beta_{1}t + \beta_{2}t^{2} + \beta_{3}t^{3} + \beta_{4}t^{4} + \beta_{5}t^{5} $, per rendere ragione della oscillazione notata col procedere dell'epidemia sui dati scalati secondo inversa della funzione logistica normalizzata: $\ln\left( \frac{y}{1-y}\right)$. Forse una spline molto smooth e con basso overfitting potrebbe essere più affidabile per le estrapolazioni. Da provare.
Non escludo di rivedere la funzione $\beta(t)$ e trovarne una più generale e/o più esplicativa, analizzando ulteriori insiemi di dati e tenendo conto di altre variabili (con opportune metriche), come la densità di popolazione, lo stile di vita diverso che può influenzare il contagio in aree diverse, ec.
Un criterio importante per scegliere le funzioni $\beta(t)$ e $i_{c}^{\ast}(t)$ è la capacità di estrarre, dall'andamento iniziale dell'epidemia, l'informazione che determina l'evolvere dell'epidemia nelle fasi successive: cioè la sua capacità previsiva.
Al momento, il modello fisico-matematico diventa perciò:
$f(t)= c_{t}^{\ast} {\frac {1}{1+e^{-(\beta_{0}
+ \beta_{1}t
+ \beta_{2}t^{2}
+ \beta_{3}t^{3}
+ \beta_{4}t^{4}
+ \beta_{5}t^{5}
)}}} $,
dove $\beta_{0} \dots \beta_{5}$ e $i_{c}^{\ast}$ sono parametri liberi oggetto di fitting.
La curva dei casi giornalieri è la derivata prima di $f(t)$:
$f_{}^{\prime}(t)= c_{t}^{\ast}
\frac{
(\beta_{1} + 2\beta_{2}t + 3\beta_{3}t^{2} + 4\beta_{4}t^{3} + 5\beta_{5}t^{4})
\cdot e^{-(\beta_{0} + \beta_{1}t + \beta_{2}t^{2} + \beta_{3}t^{3}+ \beta_{4}t^{4} + \beta_{5}t^{5})}
}
{\left( 1+e^{-(\beta_{0} + \beta_{1}t + \beta_{2}t^{2} + \beta_{3}t^{3}+ \beta_{4}t^{4} + \beta_{5}t^{5})} \right)^2}
$
Alla data di questa stima: 13-jul-2020, valgono i seguenti parametri stimati dal fitting: $$\begin{eqnarray} i_{c}^{\ast} &=& 0.008178 \nonumber \\ \beta_{0} &=& -6.445463 \nonumber \\ \beta_{1} &=& +0.340332 \nonumber \\ \beta_{2} &=& -0.007129814 \nonumber \\ \beta_{3} &=& +8.76724707e-5 \nonumber \\ \beta_{4} &=& -5.46321044e-7 \nonumber \\ \beta_{5} &=& +1.3556051e-9 \nonumber \\ R^{2}&=&0.999749 \qquad \text{(coefficiente di determinazione di } P(t) \text{ della trasformata logistica)}\nonumber \\ && \nonumber \end{eqnarray}$$
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
Picco ed estinzione si riferiscono al momento di riconoscimento/rilevamento della patologia macroscopica da parte del sistema terapeutico, con diagnosi ipoteticamente fatte nello stesso modo durante lo sviluppo della curva fino al momento del fitting. Altrimenti ulteriori distorsioni si accumulano.
Picco ed estinzione NON SI RIFERISCONO AL picco e all'estinzione dell'affollamento dei luoghi di terapia. Questi ultimi dipendono anche dalla durata del trattamento terapeutico, oltre che dall'afflusso dei manifestanti patologia macroscopica.
I dati del Ministero della Salute rappresentano la manifestazione del patologia macroscopica generatasi in assenza di azioni di contenimento del virus, almeno fino al momento in cui le azioni di contenimento, eventualmente, potranno mostrare la loro efficacia. Alla data del picco (23-mar-2020) non possono aver già mostrato effetto a causa del ritardo di fase stimabile.
I dati non rappresentano la diffusione del virus perché è largamente incognita la relazione altamente non lineare tra diffusione del virus e diffusione del patologia macroscopica. Questa relazione è influenzata da molti fattori non lineari (es: spettro della suscettibilità della popolazione al virus, spettro della reazione immunitaria della popolazione, spettro della reazione infiammatoria della popolazione, spettro dei comportamenti della popolazione, distribuzione della densità di popolazione, ec.). Inoltre il campione di soggetti intercettati dal sistema terapeutico potrebbe essere molto deviato da molti fattori: spettro individuale di percezione dei sintomi lievi, assenza di sintomi, disponibilità individuale a chiedere consiglio medico, reattività del sistema terapeutico, ec.
Dalla curva dei dati della patologia macroscopica, questo virus mostra una velocità di contagio molto più alta di quanto ricavabile dalle stime di $R_{0}$ che mi sono pervenute. Nello scritto teorico in preparazione, c'è una riflessione sulle stime di $R_{0}$. La stima diretta di $R_{0}$ (o $R_{t}$) è difficle da fare direttamente; inoltre essa è soggetta a incertezza statistica di misura molto ampia. La corretta stima di $R_{0}$ è cruciale per orientare le decisioni di azione in emergenza: contenimento diffusione virus VS potenziamento terapie d'emergenza. Se veloce, allora la terapia d'emergenza sale di punteggio. Se lento, allora il contenimento sale di punteggio. Contenimento assoluto è impossibile, e non può essere mantenuto a lungo col rigore necessario per una miriade di ragioni niente affatto futili, tanto più di fronte a un virus veloce ed efficiente a contagiare. Se veloce, e con rapido decadimento delle difese immunitarie acquisite, allora l'infezione rispunta come i funghi quando si pensa sia sotto controllo. Questo è quanto vedo teoricamente. Le sperimentazioni diranno di più. La corretta stima di $R_{0}$ può orientare meglio la valutazione della contagiosità degli asintomatici. Dubito che una asintomatico non contagi. Non ha molto senso dal punto di vista biochimico. Dipende dalla sua carica virale.
Inoltre i dati rappresentano solo quei casi di patologia macroscopica che il sistema terapeutico ha intercettato. Non rappresentano tutti quegli altri eventuali casi non diagnosticati e ignoti, ma probabilmente esistenti e guariti, o non evoluti in patologia macroscopica. Infatti il campione di soggetti intercettati dal sistema terapeutico potrebbe essere molto deviato da molti fattori: spettro individuale di percezione dei sintomi lievi, assenza di sintomi, disponibilità individuale a chiedere consiglio medico, reattività del sistema terapeutico, ec.
Inoltre ancora, i dati inglobano anche le distorsioni indotte da cambiamenti di modalità di rilevamento fatte durante il rilevamento e differenze di modalità di rilevamento da un luogo all'altro (es: criteri di pratica del tampone; tampone fatto a chi soggettivamente dichiara o
Forse un rilevamento a campione random rappresentativo di una popolazione darebbe una immagine spazio-temporale più realistica ed efficace della diffusione virale, se questo è quello che si cerca. Ma non è detto. Mentre le azioni di contenimento del virus possono servire ad ancora scarsa diffusione del virus solo per contenere l'onda di patologia macroscopica, e i tamponi ai sospetti positivi servono a orientare la terapia.
L'andamento temporale dei casi giornalieri contiene in sé $ R_{0} $ e l'andamento di $ R_{t} $. Di fatto, l'andamento della curva epidemica è l'effetto diretto della azione dell'indice di contagiosità $ R_{t} $ durante i precedenti $ g_{i} $ giorni di infettabilità.
L'idea che si propone qui è quella estrarre quella informazione e di stimare $ R_{0}$ ed $ R_{t}$ direttamente dall'andamento dei casi giornalieri derivati analiticamente dalla curva dei casi totali cumulati $f(t)$ oggetto di fitting, curva che automaticamente compensa alcuni errori nel conteggio giornaliero dei casi.
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
Dalla definizione epidemiologica: numero di infezioni secondarie causate da un singolo caso di malattia, $R_{0}$ e $R_{t}$ sono analoghi al moltiplicatore del capitale iniziale unitario dopo 1 periodo (detto anche montante), in un processo di capitalizzazione composta.
Si possono stimare $R_{0}$ o $R_{t}$ dalla curva epidemica dei casi totali introducendo il concetto di tasso di riproduzione istantaneo $R_{ist}$, analogo al tasso di capitalizzazione istantaneo in matematica attuariale.
$R_{t}$ è analogo al moltiplicatore generato dal tasso di interesse $r$ nel periodo unitario di $g_{i}\,\mathrm{giorni}$ in una capitalizzazione composta. Esempio: tasso $r=3\%$; moltiplicatore del periodo risultante $R=1.03$ per 1 periodo di $g_{i}=365\,\mathrm{giorni}$; tasso istantaneo risultante $ r=\ln(1.03)$; $ R_{ist}=\ln(1.03)/365 \approx 8.098\cdot 10^{-5} $; moltiplicatore ricalcolabile per 365 giorni (1 periodo): $e^{(8.098\cdot 10^{-5} \times 365)} = 1.03$; per 730 giorni (2 periodi): $e^{(8.098\cdot 10^{-5} \times 730)} = 1.0609$; per $n \times 365$ giorni ($n$ periodi): $e^{(n\times 365 \times 8.098\cdot 10^{-5})} = (1.03)^{n}$; ec.
$R_{ist}$ è la pendenza all'istante $t$ della curva esponenziale conseguente alla definizione epidemiologica di $R_{t}$ rappresentata in scala logaritmica che, moltiplicata per i $g_{i}$ giorni di infettività, produce il moltiplicatore $R_{t}$ dei casi all'istante $t$. $R_{0}$ è quella stessa pendenza riscontrata all'istante $t=0$ sulla curva dei casi giornalieri rappresentata in scala logarimica (in base $e$).
In questo modo, determinando $R_{ist}$ posso calcolare in ogni istante $R_{t}$ per un determinato periodo di infettività $g_{i}$ noto o convenzionale. Faccio notare che il periodo di infettività medio di un soggetto infettante in una epidemia dovrebbe variare poco: lo si può assumere costante, determinato una tantum, o determinato periodicamente, o convenzionale. Le relazioni che legano il fitting dei casi totali cumulati $f(t)$, $R_{ist}$ e $ R_{t}$ sono le seguenti:
Si può stimare $R_{ist}$ andando a vedere quando $h(t)=0$ (inizio epidemia) e ricavare $R_{ist}$ a quella data. Da questo si può pertanto stimare $R_{0}$ al limite iniziale dell'epidemia, come prevede la sua definizione epidemiologica, in $R_{0}= e^{(0.5111 \times 15)} \approx 2136.4$, assumendo un periodo di infettività di $15$ giorni; pari ad un tempo di raddoppio dei contagiati in $g_{i}=1.4 $ giorni (un giorno e mezzo).
I grafici mostrano gli strumenti per stimare $R_{0}$ e $R_{ist}$ a partire dalla curva dei casi rilevati. Secondo la definizione epidemiologica di $R_{0}$, esso ha un andamento esponenziale al limite iniziale del manifestarsi dell'epidemia. Esso evolve poi per assumere valore $R_{t}=1$ (un infetto ne infetta un altro) quando raggiunge lo stato stazionario, per poi calare ($R_{t}<1$) nella fase di estinzione dell'epidemia.
La evoluzione di $R_{0}$ evidenzia l'andamento secondo la funzione logistica di un fenomeno epidemico, determinato dalla rispettiva equazione differenziale generatrice della funzione logistica.
L'equazione differenziale generatrice della funzione logistica descrive la condizione di un agente patogeno in un ambiente a risorse rimitate che, crescendo di numero, tende ad esaurire il bacino di potenziale ulteriore espansione. L'ambiente a risorse limitate per il patogeno è costituito dal numero finito degli ospiti infettabili residui e dalla insorgente resistenza dell'ambiente (risposta immunitaria diretta della popolazione ospite e/o mutati comportamenti di ospite e/o patogeno).
Il metodo di stima di $R_{0}$ ed $R_{t}$ dalla curva dei casi totali cumulati si basa sulla determinazione dello equivalente $R_{ist}$, il quale è l'inclinazione (il coefficiente agolare) della retta tangente nel punto di interesse sulla curva dei casi giornalieri rappresentata in scala logaritmica. Rappresentare in scala logaritmica l'andamento esponenziale proprio della definizione di $R_{0}$ ed $R_{t}$ può essere pensato in scala logaritmica come una retta. Questo consente di usare il coefficiente angolare $\frac{\mathrm{d}}{\mathrm{d}t}h(t)$ della tangente alla curva dei casi giornalieri in scala logaritmica come stima di $R_{ist}$, da cui poi si ricava $R_{t}$ o $R_{0}$ moltiplicando $R_{ist} \cdot g_{i}$.
In questo modo si ottiene una stima statisticamente accurata di $R_{t}$ così come si manifesta nella realtà epidemica: di fatto è una stima di $R_{t}$ fatta mediando tutti i casi rilevati. Inoltre, con questo metodo si riesce a determinare e aggiornare $R_{t}$ nello stesso istante in cui si aggiunge un nuovo rilevamento dei casi positivi. Occorre ancora stimare direttamente $g_{i}$, ma basta accordarsi a usare sempre lo stesso per tutta la durata dell'epidemia, salvo evidenza contraria. Di fatto quello che interessa maggiormente è la proiezione del tempo di raddoppio o dimezzamento dei casi. E questo è ben rappresentato dall'andamento reale della curva epidemica.
La stima a partire da $f(t)$ -- stima derivata analiticamente dal fitting aggiornato della curva dei casi totali cumulati di un numero enorme di casi -- è una stima di $R_{t}$ statisticamente molto stabile e accurata, nonché up to date, perché media "staticamente" tutti i casi rilevati fino alla data più recente, senza l'effetto del ritardo di fase introdotto da una stima diretta di $R_{t}$ (oltre le difficoltà di stima e le incertezze statistiche di essa), o da ogni media mobile o concetto analogo di media "dinamica", tipo: filtri FIR o IIR, ec. Vedere fitting nella figura iniziale.
Va detto che, con il procedere dell'epidemia, si sono sommati come nuovi casi anche accertamenti ripetuti sullo stesso soggetto. Un data set deputato dai conteggi plurimi sullo stesso soggetto avrebbe consentito un fitting $f(t)$ più accurato e calzante sul fenomeno. Meglio ancora se si fossero anche determinati i casi univoci sempre con lo stesso metodo.
Casi:
Dettagli, dimostrazioni ed esempi del metodo nello scritto esteso linkato a fine pagina.
Meccanismi evolutivi fondamentali evidenziano che se un patogeno parassita è altamente letale per l'ospite, allora tende a diffondersi con difficoltà, in quanto tende a uccidere il suo ospite e di conseguenza a causare la propria difficoltà a diffondersi.
Viceversa, un patogeno parassita tendenzialmente innocuo per l'ospite può facilmente evolvere per avere alta e veloce diffusività, avvantaggiandosi sui suoi competitors meno veloci e meno inclini a diffondersi, a pari innocuità per l'ospite e a pari mobilità della popolazione degli ospiti. Non nuocere all'ospite genera un ambiente favorevole al patogeno più veloce e incline a diffondersi proprio perché non nuoce all'ospite.
Un virus non nasce dal nulla. Arriva da altri serbatoi in cui è evoluto. E per evolvere non li ha distrutti. Virus e batteri sono lì da alcuni miliardi di anni.
La distanza evolutiva tra ominidi e pipistrelli (o altro serbatoio di mammiferi) è molto piccola, se vista dal punto di vista del virus. Perciò un antenato prossimo di questo virus, così efficiente e veloce a contagiare, non può essere stato troppo nocivo per quei mammiferi, pena la sua estinzione conseguente. Molto improbabile che possa esserlo per noi.
Se si ragiona in termini evoluzionisti, la strategia del virus è molto sensata. O, meglio: il meccanismo di propagazione di questo virus mostra essere molto efficace, dato che attribuire intenzionalità al meccanismo evolutivo è profondamente sbagliato: il processo evolutivo si limita ad esplorare per tentativi il possibile chimico-fisico, tentativi setacciati dall'idoneità contingente all'ambiente.
Manifestarsi in patologia macroscopica con forte ritardo di fase --- insieme a infettare velocemente; insieme a fare pochi danni sull'ospite, soprattutto se giovane e femmina, cioè in modo tale da non danneggiare la potenzialità riproduttiva dell'ospite, dato che, almeno tra i mammiferi, non servono molti maschi per popolare un ambiente, mentre femmine sì; insieme a una probabile scarsa impronta a rapido decadimento sul sistema immunitario dell'ospite --- pare una strategia di successo, dal punto di vista del virus. Ed è probabilmente quello che succede nel serbatoio animale da cui si pensa provenga il Corona Virus di COVID-19. In fondo, dal punto di vista del virus, i pipistrelli non sono molto distanti dagli ominidi, come ambiente da colonizzare.
Tutto ciò avviene per fondamentali caratteristiche del meccanismo evolutivo scoperto da Charles Darwin e ulteriormente sviluppato dal pensiero evoluzionista: Ernst Mayr, Richard Dawkins, Daniel Dennett (per gli aspetti teorico/algoritmici), e molti altri notevoli pensatori.
Se l'epidemia evolve come sta tratteggiando il modello logistico (rapidità di contagio latente e infettante con scarsa impronta sull'ospite), avremo una ulteriore conferma di questa inferenza evoluzionista.
In ogni caso va tenuto a mente che nulla si può mai escludere in universo chimico-fisico indifferente alla sofferenza dei suoi prodotti, quali noi siamo. La cautela è d'obbligo. Così Charles Darwin ad Asa Gray:
"Mi sembra che ci sia troppa sofferenza nel mondo. Non riesco a persuadermi che un Dio benefico e onnipotente avrebbe potuto creare gli Ichneumonidæ con l'intenzione esplicita di farli alimentare dei corpi viventi dei bruchi dall'interno, o che un gatto debba giocare con i topi."
https://www.darwinproject.ac.uk/letter/DCP-LETT-2814.xml
Per approfondire l'aspetto evoluzionistico in proposito, è molto interessante l'articolo di Gilberto Corbellini, La pandemia è neodarwiniana, e i virus mancano di buon senso.
Comunque, da quanto si intravvede, questo virus non sembra l'avvento dell'Apocalisse. Quel genere letterario è molto amato. Infatti l'onda montante degli Apocalittici è in fermento.
Nei grafici di questa sezione si mostra l'efficacia ipotetica di un lockdown che riduca al 20% l'insorgenza di nuovi contagi rispetto a quanto misurato su una epidemia che si espande naturalmente senza modifiche nel comportamento degli infettati/infettanti, cioè come si evidenzia prima del lockdown.
I grafici sotto mostrano l'efficacia ipotetica di un lockdown che abbia azione immediata. In realtà un lockdown avrà una efficacia differita nel tempo per un tempo di latenza dell'infezione virale al momento ignota. In ogni caso, se il lockdown avesse avuto effetto, essendo una azione entrata in vigore quasi istantaneamente, si dovrebbe notare, da qualche parte sulla curva dell'andamento epidemico, una discontinuità simile a quella evidenziata nei grafici. Invece non si vede.
La curvatura nel grafico scalato secondo inversa della funzione logistica -- grafico che proietta la curva epidemica verso una inclinazione meno ripida -- comincia a mostrarsi prima che il lockdown possa aver avuto effetto. In altre parole, la curva epidemica sembra non spostarsi dal suo andamento naturale determinato dall'andamento precedente il lockdown. La cosa è più evidente col modello aggiornato che adotta una funzione non lineare per il fitting e che fitta meglio della ipotesi lineare del modello iniziale. Studierò meglio questo aspetto.
L'eventuale effetto del lockdown allontana l'estinzione dell'epidemia.
Il fatto che l'effetto del lockdown si mostri poco fa sorgere alcune domande fondamentali:
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
| -20 | -10 | 0 | 10 | 20 | 30 | 40 | 50 | 60 |
| 04-Feb-2020 | 14-Feb-2020 | 24-Feb-2020 | 05-Mar-2020 | 15-Mar-2020 | 25-Mar-2020 | 04-Apr-2020 | 14-Apr-2020 | 24-Apr-2020 |
| 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 |
| 04-May-2020 | 14-May-2020 | 24-May-2020 | 03-Jun-2020 | 13-Jun-2020 | 23-Jun-2020 | 03-Jul-2020 | 13-Jul-2020 | 23-Jul-2020 |
Ho preparato una tabella che mette a confronto le incidenze di casi e morti a confronto. La cosa più evidente che qualitativamente emerge è la correlazione tra diffusione VS vivere in una metropoli dei paesi ricchi.
Si direbbe una correlazione con lo stile di vita che si pratica nelle metropoli dei paesi ricchi: alta densità di popolazione; la popolazione che ha uno stile di vita metropolitano specifico (trasporto collettivo; vita al chiuso con ventilazione forzata in ambienti affollati: ufficio, palestra, cinema, ristorante, ec.; tipologie edilizie con frequente ventilazione forzata; vita di relazione intensa; ec.)
Tabella delle incidenze di contagiati e morti in alcuni paesi.
In questo sito si trovano tutti i dati per capire meglio i confronti tra nazioni diverese: https://ourworldindata.org/Alcune premesse per inquadrare la rilevanza e la mortalità di COVID-19 sulla "fisiologia" di una popolazione sono d’obbligo.
Questo non cancella il fatto che la necessità inattesa e incognita di extra terapie intensive e sub-intensive, di ausili respiratori, farmaci e altro, in regime di alta e veloce contagiosità specifica del virus e pandemica, causi stress al limite della tenuta del sistema terapeutico di un paese. Inoltre il numero di quanti vanno a sviluppare il patologia macroscopica, stimabile sulla base del metodo attuale di identificazione COVID-19, sono imponenti e concentrate nel tempo. Le conseguenze sul sistema terapeutico sono immediatamente evidenti e pesanti.
Ecco le premesse:
Per esempio:
Considerazioni più estese nello scritto esteso linkato a fine pagina.
I am tinkering with a logistic regression model of COVID-19 cumulative count of macroscopic disease bearing cases in Italy and its underlying equations. It fits well ($R^{2}>0.99974$). Some theoretical considerations are being drawn, mainly on $R_{0}$ estimate based on curve shape of measured data and the very low contagion ratio $i_{c}^\ast$ with a high $R_{0}$.
$R_{0}$ appears astonishingly high, according to equations: $R_{0}= 2.0 \parallel 1.4~\mathrm{days}$, that is doubling the population of infected among infectables in 1.4 days
If it is really so, then the virus appears to be difficult to take under control by actions on population behaviour because of possible latency of the virus and phase lag of the macroscopic disease. Moreover, it shall pop up again, like mushrooms, if only acquired immunity will show rapid decay. Very likely, we shall have to learn to cohabit with it in the same way we live with other deseases with high $R_{0}$. Also, the macroscopic disease caused by the virus should hit only a minimal and selectively fragile/susceptible fraction of the population. Possible causes of this may be a population partially protected by unknown or not yet hypothesised mechanisms, by undetecting asyntomatic but infecting subjects, by unknown environmental factors or susceptibility spectrum, or by a combinations of these things. This can be seen by wisely perusing the theoretical model drawn here. Anyway, caution on theoretical models of unknown threats is mandatory. We'll see...!
[ This link is also achived on: https://web.archive.org ]
[ Questo link è anche archiviato su: https://web.archive.org ]
Abstract
Si è costruito un semplice modello parametrico basato sulla funzione logistica per tentare di descrivere l'andamento temporale dell'epidemia COVID-19 in Italia, usando i dati ufficiali del Ministero della Salute italiano. Il modello evidenzia anomalie nell'indice di contagio, rispetto a quanto ci si potrebbe aspettare da un contagio che si diffonde così velocemente in una popolazione presunta vergine al virus. Interrogativi sulla velocità di contagio e sulle stime di $ R_{0} $ vengono evidenziati ed esposti. Considerazioni vengono esposte sulla base delle evidenze del fitting parametrico. Dubbi vengono esposti sulla efficacia delle azioni di contenimento della diffusione del virus a causa della velocità di contagio, almeno per Lombardia e zone limitrofe. L'epidemia potrebbe esaurirsi prima che le azioni di contenimento alla diffusione del virus possano diventare efficaci. Cautela va comunque usata nel fidarsi di modelli matematici di fenomeni molto complessi e abbastanza ignoti.
<work in progress>